- ZedIoT
-
-
-
血常规检查是医学诊断中最常见的检查项目之一,它能够提供有关患者健康状况的重要信息。然而,血常规细胞的手动标注和计数工作繁琐且易受人为因素影响。因此,基于深度学习技术的自动标注和细胞计数系统可以显著提高工作效率和准确性。本文将详细介绍如何通过深度学习技术实现血常规细胞的自动标注、训练模型,并进行细胞数量识别的技术方案。
数据收集与标注
1. 数据收集
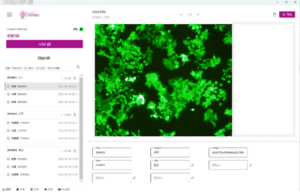
首先需要从医院或研究机构收集大量的血常规细胞图像数据。这些数据应包括不同类型的细胞,如红细胞、白细胞和血小板,并尽可能覆盖各种病理状态,以提高模型的泛化能力。
2. 数据标注
为了训练深度学习模型,必须对血常规细胞图像中的细胞进行精确的标注。常用的开源标注工具包括:
● LabelImg:支持创建 PascalVOC 和 YOLO 格式的标注文件,适合简单的矩形标注任务。
● LabelMe:支持多种标注类型,包括多边形,适合复杂的标注任务。
● CVAT (Computer Vision Annotation Tool):功能强大,适合大规模数据的标注,支持视频帧的标注。
标注流程
- 导入图像数据:将血常规细胞图像数据导入标注工具。
- 标注细胞:对每张图像中的细胞进行标注,记录其位置和类别。
- 保存标注文件:将标注结果保存为指定格式文件,供模型训练使用。
数据预处理
1. 图像预处理
为了提高模型的性能,对图像进行预处理是必要的步骤。常见的预处理方法包括灰度化、归一化和直方图均衡化等。
import cv2
def preprocess_image(image_path):
image = cv2.imread(image_path, cv2.IMREAD_GRAYSCALE) # 灰度化
image = cv2.equalizeHist(image) # 直方图均衡化
image = image / 255.0 # 归一化
return image2. 数据增强
数据增强是提升模型性能的重要手段。常用方法包括随机旋转、水平和垂直翻转、随机裁剪和颜色抖动。可以使用 TensorFlow 或 PyTorch 提供的数据增强工具,也可以使用 albumentations 等第三方库。
import albumentations as A
from PIL import Image
import numpy as np
def augment_image(image):
transform = A.Compose([
A.HorizontalFlip(p=0.5),
A.RandomBrightnessContrast(p=0.2),
A.Rotate(limit=20, p=0.5)
])
augmented = transform(image=np.array(image))
return Image.fromarray(augmented['image'])模型设计与训练
1. 模型选择
根据任务需求选择适合的深度学习模型:
● CNN(卷积神经网络):适合图像分类和特征提取。
● U-Net:适合图像分割任务,可以精确定位细胞位置。
● YOLO(You Only Look Once):适合实时目标检测任务,能够在一张图像中快速检测多个细胞。
● Faster R-CNN:在目标检测任务中表现出色,适合精确的细胞检测。
2. 模型训练
以 YOLO 为例,使用 TensorFlow 进行模型训练:
import tensorflow as tf
from yolov3_tf2.models import YoloV3
from yolov3_tf2.dataset import transform_images, load_tfrecord_dataset
from yolov3_tf2.utils import load_darknet_weights
# 加载数据集
train_dataset = load_tfrecord_dataset('path/to/train.tfrecord', 'path/to/classes.txt')
train_dataset = train_dataset.map(lambda x, y: (transform_images(x, 416), y))
# 加载模型
yolo = YoloV3(classes=NUM_CLASSES)
load_darknet_weights(yolo, 'path/to/yolov3.weights')
# 编译模型
yolo.compile(optimizer=tf.keras.optimizers.Adam(),
loss=[tf.keras.losses.CategoricalCrossentropy(from_logits=True),
tf.keras.losses.BinaryCrossentropy()],
metrics=['accuracy'])
# 训练模型
yolo.fit(train_dataset, epochs=EPOCHS, steps_per_epoch=STEPS_PER_EPOCH)
# 保存模型
yolo.save_weights('path/to/yolov3_weights.h5')3. 模型评估与优化
模型训练完成后,需要对其进行评估。常用评估指标包括准确率、召回率和 F1-score。通过调整模型结构、增加数据量以及使用更先进的优化算法,可以进一步提高模型性能。
# 评估模型
eval_dataset = load_tfrecord_dataset('path/to/eval.tfrecord', 'path/to/classes.txt')
eval_dataset = eval_dataset.map(lambda x, y: (transform_images(x, 416), y))
yolo.evaluate(eval_dataset)系统集成与部署
1. 实时图像处理
为了在实际应用中实现实时细胞识别,需要将训练好的模型集成到图像处理系统中。可以使用 OpenCV 处理实时图像,并将每一张图像传递给模型进行预测。
2. 系统架构
一个典型的实时细胞识别系统架构包括以下组件:
● 图像采集模块:负责采集血常规细胞图像。
● 图像处理模块:负责图像的预处理。
● 细胞识别模块:加载训练好的深度学习模型,对每一张图像进行细胞识别和计数。
● 结果展示模块:将识别结果实时显示,并生成报告。
3. 部署
系统部署需要考虑计算资源和响应时间。可以选择在本地服务器或云端部署模型,并利用 GPU 提高计算效率。
以下是一个简单的实时细胞识别示例代码:
import cv2
import tensorflow as tf
from yolov3_tf2.models import YoloV3
from yolov3_tf2.dataset import transform_images
# 加载模型
yolo = YoloV3(classes=NUM_CLASSES)
yolo.load_weights('path/to/yolov3_weights.h5')
# 打开图像
image_path = 'path/to/image.jpg'
image = cv2.imread(image_path)
# 预处理图像
image_processed = transform_images(image, 416)
# 进行细胞识别
boxes, scores, classes, nums = yolo(image_processed)
# 在图像上绘制识别结果
for i in range(nums[0]):
x1, y1, x2, y2 = boxes[0][i]
cv2.rectangle(image, (x1, y1), (x2, y2), (0, 255, 0), 2)
cv2.putText(image, f'{classes[0][i]}: {scores[0][i]:.2f}',
(x1, y1 - 10), cv2.FONT_HERSHEY_SIMPLEX, 0.9, (36,255,12), 2)
# 显示结果
cv2.imshow('Image', image)
cv2.waitKey(0)
cv2.destroyAllWindows()本文介绍了基于深度学习的血常规细胞自动标注和数量识别技术方案。从数据收集与标注、数据预处理、模型设计与训练,到系统集成与部署,详细描述了每一步的技术细节。通过该方案,可以构建一个高效的细胞识别和计数系统,提升血常规检查的效率和准确性。然而,在实际应用中,仍需不断优化模型和系统,以应对复杂多变的临床环境。未来,随着深度学习技术的不断进步,自动化细胞识别系统将在医学诊断领域发挥越来越重要的作用。
典型应用介绍